Visor de glosario
Pequeño diccionario de Técnicas de Laboratorio y Modelos Animales
- A
- Aequorea victoria
Aequorea Victoria es un tipo de medusa bioluminiscente del orden Hydroida.
Descripción
Es conocida como “gelatina cristal” debido a su transparencia.
Consta de una boca grande y tentáculos fuertes fáciles de contraerse, además, estos tentáculos poseen una toxina con la que atrapan a sus presas.
El diseño acampanado de su cuerpo le permite moverse con una mayor facilidad por las aguas a diferencencia de otro tipo de medusas. Esta campana puede alcanzar un diametro de 25 cm,pero usualmente no mide más de 8 cm.

Hábitat
Auqeueroa victoria se localiza en la costa occidental de Norteamérica hasta el sur de California. Aunque suelen situarse alrededor de la costa, también podemos encontrarlas mar adentro debido a corrientes marinas o al viento.
Decimos que son medusas estacionales ya que desaparecen totalmente del agua en otoño y vuelve a parecer en primavera donde empeziezan el ciclo de nuevo.

Alimentación
Se alimenta de pequeños organismos blandos y crustáceos,aunque prefiere alimentarse de otras medusas gracias a la capacidad de dilatar su boca la mitad de su tamaño.
Las medusas se comen a la presa entera al carecer de dientes por ello su estomago contiene una enzima que se encarga en descomponer el alimento en sustancias nutritivas.

Reproducción
No requieren una cierta época del año pero si se ven limitadas por factores como la escasez de alimento,abundancia o temperatura del agua.Aequorea Victoria se reproduce de manera sexual y asexual,es decir,los dos pueden crear óvlos y espermatozoides.

Curiosidades
Este tipo de medusa posee biolumniscencia. Esta se produce al interacionar una proteina llamada aecuorina con los iones calcio. A continuación,otra proteina verde florescente (GFP) capta la energia de la aecuorina y la libera en forma de luz verde.
A continuacion tenéis un video donde se muestra este fenómeno:
Este tipo de medusa es cosechada por su aecuorina bioluminosa pues es útil para experimentos neurológicos y biológicos para detectar el calcio.
Bibliografía:
Para saber más:
Belén Resta (2º de Bachillerato)- Ajolote
-
El ajolote (Ambystoma Mexicanum) es un anfibio, concretamente una salamandra, endémico de México, cuyo nombre científico hace referencia al nombre del dios azteca de la muerte, la resurrección y el juego.

En la actualidad, el ajolote está siendo estudiado por la ciencia debido a que se le atribuyen capacidades regenerativas extraordinarias, como el hecho de poder recuperar una extremidad de su cuerpo que hubiesen perdido anteriormente, u órganos vitales (como el corazón, la retina o el hígado) que hubiesen dejado de funcionar correctamente. Esta cualidad, tras una serie de análisis e investigaciones, podría ayudarnos a encontrar la forma de que los seres humanos también pudiésemos regenerar partes de nuestro organismo.
Relación entre la regeneración de los ajolotes y la de las falanges humanas: http://luistatlav3.wordpress.com/2009/08/08/el-ajolote-y-la-regeneracion-de-las-extremidades/
Marina Robleño Vélez
- Arabidopsis thaliana
Arabidopsis thaliana.
Planta herbácea crucífera (angiospermas dicotiledóneas) distribuida por todos los continentes que se comenzó a investigar hace 40 años. Fue descubierta por el botánico Johannes Thal en el siglo XVI, es destacada por su sencillez y facilidad de manipulación en el laboratorio, además de por el pequeño tamaño de su genoma, que se secuenció totalmente en el año 2000. Otro factor favorecedor para su estudio es su abundante producción de semillas, las cuales tienen una estructura simple y un ciclo de dispersión corto, por lo que se sitúan cerca de la planta en la germinación.

Sus flores son pequeñas y hermafroditas, blancas y agrupadas en grupos. Cuentan con un fruto con alrededor de treinta semillas. Comparte familia con especies como la col, el nabo y la rúcula. Su ciclo de vida es corto, menor a un año.


Esta especie es de las más utilizadas en los laboratorios de investigación de plantas. Se investiga la respuesta de esta a distintos niveles de estrés, su germinación o su floración. Gracias al reducido tamaño de su genoma, este puede ser manipulado mediante ingeniería genética con gran facilidad.
Su estudio nos ha permitido avanzar en el campo biológico de la floración. Sus flores están formadas por cuatro verticilos concéntricos con los cuatro órganos florales dispuestos de afuera hacia adentro: 4 sépalos (verdes), 4 pétalos (blancos), 6 estambres (órganos masculinos) y dos carpelos (órganos femeninos). Se han estudiado factores que implican cuando la planta sabe que es tiempo de florecer, por ejemplo. La planta detecta, gracias a unos fotorreceptores de sus hojas, la duración del día. La floración se acelera cuando el día se hace más largo. Cuando la planta se encuentra en unas condiciones favorables, segrega moléculas promotoras hacia el tallo para inducir la formación de las flores. Es un proceso delicado en el que actúan distintos genes.
Sus genes también nos aportan información sobre la identidad de la planta, que se puede estudiar mediante el modelo ABC. La actividad A nos informa del desarrollo de los sépalos, la B inducen la formación de pétalos y la C induce el desarrollo de estambres.
También es utilizada en el estudio de los distintos niveles de estrés (frío, salinidad…) a los que se somete la planta y su respuesta ante estos.
Enlace de vídeo, laboratorio de investigación de Arabidopsis Thaliana.
BIBLIOGRAFÍA:
- http://biologia.laguia2000.com/botanica/arabidopsis-thaliana
- http://www.devbio.biology.gatech.edu/?page_id=11214
- http://seresmodelicos.csic.es/planta.html
- http://www.uab.cat/web/detalle-noticia/arabidopsis-thaliana-no-solo-una-planta-de-laboratorio-importancia-de-las-poblaciones-silvestres-catalanas-1345680342040.html?noticiaid=1345695163636
- https://www.arabidopsis.org/
- https://www.arabidopsis.org/portals/education/aboutarabidopsis.jsp
- http://herbarivirtual.uib.es/cas-uv/especie/6202.html
- https://www.researchgate.net/publication/260076781_Arabidopsis_thaliana_como_organismo_modelo_en_Biologia
- http://cordis.europa.eu/news/rcn/15996_es.html
Autora: Celia López Pastor.(2º Bachillerato)
- B
- C
- CRISPR/Cas
CRISPR son las siglas inglesas de "Clustered Regularly Interspaced Short Palindromic Repeats" o traducido al español: “Repeticiones Palindrómicas Cortas Agrupadas y Regularmente interespaciadas.” Resumiendo, cuando hablamos de CRISPR hablamos de una molécula de ARN un poco especial. El segundo elemento del conjunto, Cas9 (CRISPR associated system), es una enzima que es capaz de cortar ADN de forma muy precisa.
Combinando estos dos elementos, obtenemos una herramienta que permite modificar secuencias de ADN cortando el genoma y con la posibiidad de añadir la variación deseada.
https://i.ytimg.com/vi/2pp17E4E-O8/maxresdefault.jpg
Este proceso consta de varios pasos:
Primero, se aisla una molécula de ARN guía que se acoplará a la enzima CAS9 y que la guiará hasta el gen defectuoso o que se quiere sustituir.
Posteriormente, la enzima Cas9 cortará el gen que se le haya designado, inutilizando o dando lugar a nuevas mutaciones del gen. En este momento, cuando la enzima CAS9 corta el ADN, se queda un hueco en la secuencia que puede rellenarse añadiendo un gen sano o que provoque la mutación perseguida.
Pinche aquí para una descripción gráfica del proceso
Este innovador método lleva desde 2012 desarrollándose, pero todo empezó en 1987 cuando se descubrió el sistema inmune de unas bacterias llamadas Streptococcus pyogenes. Estas bacterias poseen unas enzimas que son capaces de detectar ADN vírico. Cuando lo detectan, lo cortan y este ADN vírico queda inutilizado. Además de destruir la amenaza, estas bacterias cogen parte del ADN vírico y lo incluyen en su genoma. De esta manera consigue que su descendencia pueda reaccionar mucho más rápido ante estos virus.(1)

La posibilidad de modificar el genoma con la precisión que esta herramienta aporta abre las puertas a infinitas aplicaciones. Imitando el sistema inmune del estreptococos antes mencionado, podemos crear nuestros propios sistmas inmunes. Por ejemplo, se ha implantado la técnica con CRISPR en lecherías industriales ya que hay un gran problema con ataques bacteriófagos. Ademas de esto, se puededen crear animales y plantas transgénicos con mutaciones muy controladas, e incluso, modificar humanos. Este último apartado ha sido discutido y prohibido en grandes órganos políticos como la Casa Blanca debido a los problemas éticos que plantea. Aún así, en China se ha modificado con esta técnica un embrión humano con la excusa de que finalmente era inviable. Esto podría alarmarnos bastante pero si se utiliza de forma controlada puede ser de gran ayuda para el futuro de la humanidad.(2)(3)
Recientemente se han creado monos transgénicos con un genoma mucho más parecido al humano. Con esto se pretende el estudio de la terapia génica con CRISPR/CAS9 para su posterior uso en humanos. Esta técnica tiene expectativas muy altas como la cura del cáncer, de enfermedades degeneratias como el alzheimer. Resumiendo, como ha dicho el propietario de la empresa que "creó" estos monos, se pretende encontrar la cura para enfermedades graves que actualmente no son tratables.(4)(5)

Hay que darle el reconocimiento que se merece a Francisco Juan Martínez Mojica, actual candidato a los premios Nobel por el descubrimiento de este "copia y pega" genético que sin duda está revolucionando el mundo. Pinche aquí para acceder al vídeo en el que Francisco Juan Martínez Mojica explica su descbrimiento personalmente
BIBLIOGRAFÍA
-http://dciencia.es/que-es-la-tecnologia-crispr-cas9/
-http://www.news-medical.net/life-sciences/CPISPR-applications-(Spanish).aspx
-https://www.technologyreview.es/biomedicina/44296/un-nuevo-copypaste-genetico-permite-la-terapia/
-http://elpais.com/elpais/2016/09/30/ciencia/1475261168_399307.html
Autor: Eduardo Román (2º Bachillerato)- Cenaerhabditis elegans
- - El Cenaerhabditis elegans (nombre científico) es un gusano diminuto de poco más de 1mm de longitud que vive en el suelo en ambientes templados. Desde hace más de 40 años también vive en las paredes del laboratorio.- Es un organismo pluricelular en forma de tubo alargado que se adelgaza en los extremos. Todo el cuerpo está recubierto por una fina cutícula exterior. Las células están organizadas formando órganos y sistemas bastante simples.- Es un animal invertebrado que pertenece a la clase de los nematodos o gusanos de cuerpo cilíndrico y sin segmentar. Dentro del mismo grupo, hay especies terrestres y acuáticas de vida libre.- Se alimenta de microorganismos y micronutrientes y en ocasiones debe soportar períodos donde el alimento es escaso.- Tiene un sistema digestivo formado por estoma (o boca), faringe e intestino. También dispone de órganos sexuales (gónadas) y un rudimentario sistema nervioso.- A pesar de no presentar ojos tiene cierta capacidad para percibir intensidades luminosas. Su cuerpo es transparente, lo que permite visualizar con técnicas de microscopía diferentes procesos biológicos.
 En las últimas décadas, ha alcanzado el prestigio de organismos de mayor tradición, como la mosca del vinagre o el ratón, y ha sido utilizado para estudiar la genética del desarrollo y el sistema nervioso. Últimamente, también está haciendo aportaciones en el conocimiento de las causas del envejecimiento, de la muerte celular y de la estructura del genoma.El siguiente vídeo nos puede ayudar a ampliar los conocimientos sobre el Cenaerhabditis eleganshttp://www.dailymotion.com/video/x72v4a_c-elegans-un-gusano-nobel_schoolPara más información se pueden consultar las siguientes páginas:http://www.seresmodelicos.csic.es/cuc.htmlhttp://helvia.uco.es/xmlui/handle/10396/5533Pilar Romero Martín
En las últimas décadas, ha alcanzado el prestigio de organismos de mayor tradición, como la mosca del vinagre o el ratón, y ha sido utilizado para estudiar la genética del desarrollo y el sistema nervioso. Últimamente, también está haciendo aportaciones en el conocimiento de las causas del envejecimiento, de la muerte celular y de la estructura del genoma.El siguiente vídeo nos puede ayudar a ampliar los conocimientos sobre el Cenaerhabditis eleganshttp://www.dailymotion.com/video/x72v4a_c-elegans-un-gusano-nobel_schoolPara más información se pueden consultar las siguientes páginas:http://www.seresmodelicos.csic.es/cuc.htmlhttp://helvia.uco.es/xmlui/handle/10396/5533Pilar Romero Martín
- Centrifugación
¿Qué es?
La centrifugación es un método por el cual se pueden separar sólidos de líquidos de diferente densidad por medio de una fuerza centrífuga. Esta técnica puede usarse cuando la sedimentación es muy lenta para acelerar el poceso. Los componentes más pesados se sedimentan rápidamente y los livianos quedan encima.
La centrifugación se puede llevar a cabo a escala preparativa o escala analítica. La primera se utiliza para aislar partículas para su aprovechamiento posterior y la segunda permite determinar propiedades físicas como la velocidad de sedimentación o el peso molecular.
 Imagen obtenida de Procesos bio
Imagen obtenida de Procesos bioDentro de la centrifugación, hay distintas modalidades dependiendo de diversos factores:
Según su velocidad:
- Centrifugación a baja velocidad: menos de 10000 rpm.
- Centrifugación a alta velocidad: entre 10000 y 20000 rpm.
- Ultracentrifugación: más de 20000 rpm.
Dependiendo del propósito:
- Centrifugación analítica: permite determinar propiedades físicas como la velocidad de sedimentación o el peso molecular.
- Centrifugación preparativa: aislar partículas para su posterior aprovechamiento.
Según el medio en el que se centrifuga:
- Centrifugación diferencial: es el proceso que tiene como resultado la obtención de un sobrenadante y un material sedimentado.
- Centrifugación de velocidad de sedimentación: la muestra a analizar se deposita en la parte superior de un gradiente de densidades previamente formado, a causa de la fuerza centrífuga las partículas se mueven a velocidades que dependen de la masa y sedimentan en diferentes zonas del gradiente. La densidad máxima del gradiente no ha de exceder a la de las partículas a separar.
- Centrifugación de equilibrio de sedimentación: separa las partículas en un gradiente de densidades en función de la densidad de las mismas, la sedimentación final no se produce si se controlan las condiciones de centrifugación, ya que las partículas flotan sobre un "colchón" de material que posee una densidad superior a la de éstas.
- Método de barrera: se trata de una centrifugación a través de un medio de densidad constante, la densidad debe ser intermedia entre la de los tipos celulares que se quieren separar.
¿Cómo se hace?
La centrifugación en el laboratorio se realiza por medio de un aparato llamado centrífuga, en el cual se colocan tubos de ensayo que contienen la mezcla; la centrífuga gira con tal velocidad que separa el sólido y lo deposita en el fondo del tubo.
Las centrífugas permiten someter a las muestras a intensas fuerzas que producen la sedimentación en poco tiempo.En las centrífugas se controla la temperatura de la cámara para evitar el sobrecalentamiento debido a la fricción. Existen tres tipos:
- Centrífugas (hasta 3000 gr.)
- Súper-centrífugas (2000-20000 gr.)
- Ultracentrífugas (15000-600000 gr.)
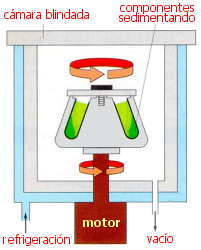
Imagen de Biomodel
En este video que adjunto se muestra como es la técnica de la centrifugación:
Para ampliar la información sobre esta técnica, les dejo la siguiente referencia sobre la web (Biomodeluahes, 2016)
WEBGRAFÍA
Autora: Sofía Ferrer (2º de Bachillerato)- Chip de ADN
¿Qué son?
Los Chips de ADN, también denominados microarrays, es una técnica basada en realizar análisis genéticos, con el fin de comparar las expresiones génicas producidas en dos situaciones diferentes, por lo que se estudia miles de genes de forma paralela. Por ejemplo, la expresión en unas células normales y en otras células cancerígenas. 1
Sin embargo, los datos generados por esta herramienta, son muy variables y de gran volumen, por lo que son imprescindibles tanto en el diseño experimental como en el análisis estadístico, con el objetivo primordial de tratar los diversos problemas biológicos. 2

Imagen 1: Microarrays con 40 000 sondas. Fuente: wikipedia
Funcionamiento y preparación
El funcionamiento de esta técnica está basada en la hibridación del ADN, es decir, la unión de dos hebras de ADN complementario. De este modo, se utiliza pequeñas proporciones de ADN, pertenecientes varios genes, cuya expresión quiere ser medida y estudiada. Para ello, se necesitan robots, que utilizan agujas especiales para obtener las moléculas y colocarlas en una base de cristal. Estas muestras se denominan dianas. 3
De las células que se quieren medir su expresión génica, se obtienen una muestra de ARN, que pasará a ser ADN complementario (ADNc), mediante una transcriptasa reversa, y en este proceso se marca con una molécula fluorescente, fluorcromo, utilizando uno distinto para cada muestra. A esta prueba marcada se le denomina sonda, y tendrá que enfrentarse a las dianas del microarray. 4
Cada partícula de ADN complementario marcada de la sonda anterior, comenzará un movimiento por difusión hacia la diana que incluya su otra molécula complementaria a esa hebra de ADN, para así, hibridarse con ella, y fijarse a su estructura. A continuación, se deja reposar la muestra con el fin de que las moléculas complementarias se hibriden. Tras esto, se procede a lavar el microarray, para así, medir la cantidad de ADN de la sonda de cada diana. 5
Finalmente, un escáner láser obtiene dos imágenes, una para cada fluorcromo, donde se puede observar distintos puntos de luz, cuyas intensidades difieren en el nivel de hibridación en cada diana. 6

Imagen 2: Hibridación. Fuente: Introduction to DNA microarrays
Fuente vídeo: Youtubecom. 2016. YouTube. [Online]. [8 October 2016]. Available from: https://www.youtube.com/watch?v=VNsThMNjKhM
Análisis de la imagen obtenida
El análisis de la imagen es un proceso fundamental ya que determina los valores que serán analizados en el estudio estadístico. El proceso de análisis de la imagen se divide en tres etapas:
- Localización de los puntos proporcionados por el fabricante del microarray.
- Determinar el número de puntos, y conocer su respectiva agrupación.
- Segmentación: identificar que píxeles pertenecen a un punto, y cuales son píxeles de fondo.
- Cuantificación: se analiza la intensidad de la señal, es decir. se realiza el promedio o la mediana de las intensidades de los píxeles que constituyen un punto. 6

Imagen 3: Análisis de la imagen. Fuente: Aspectostecnicos
Aplicaciones
La primera aplicación de microarrays consistió en medir paralelamente el nivel de expresión génica de miles de genes. Pero los avances tecnológicos han mejorado su calidad, ampliando sus aplicaciones en el mundo de la medicina, sobre todo en las investigaciones genéticas. 7
En actualidad, los chips de ADN sirven para estudiar cualquier problema biológico:
- Detección de mutaciones
- Predecir la respuesta a un tratamiento
- Identificar genes específicos a una patología
- Diagnosticar enfermedades infecciosas
- Estudiar genes que se expresan de forma diferentes ante varias condiciones. Por ejemplo: sanos y enfermos.
- Permite realizar estudios de diagnóstico de tumores, gracias a la detección de genes expresados en un tejido determinado.
- Detectar genes relacionados con la quimioresistencia. 8,9
Observe esta animación para entender mejor esta técnica. 10
Página web de interés: Genome
Bibliografía
- Blogspotcomes. 2016. Blogspotcomes. [Online]. [29 September 2016]. Available from: http://noticiasbiociencias.blogspot.com.es/2011/12/nociones-de-biologia-que-son-los.html
- Scehues. 2016. Scehues. [Online]. [29 September 2016]. Available from: http://www.sc.ehu.es/ccwbayes/docencia/mmcc/docs/divulgativos/UsoDeChipsDeADN.pdf
- Blogspotcomes. 2016. Blogspotcomes. [Online]. [29 September 2016]. Available from: http://morfoudec.blogspot.com.es/2008/08/chip-de-adn.html
- Seioes. 2016. Seioes. [Online]. [29 September 2016]. Available from: http://www.seio.es/BEIO/files/BEIOVol22Num2_AE_MJRivas JMSanchez JRivas.pdf
- Bitesizebiocom. 2011. Bitesize Bio. [Online]. [29 September 2016]. Available from: http://bitesizebio.com/7206/introduction-to-dna-microarrays/
- Drosophilaes. 2012. Boletin drosophila. [Online]. [29 September 2016]. Available from: http://www.drosophila.es/blog/2012/04/09/chips-de-adn-o-microarray/
- Personalesupves. 2016. Personalesupves. [Online]. [29 September 2016]. Available from: http://personales.upv.es/jcanizar/modulo_3/aspectos_tecnicos_1.html
- Elsevieres. 2016. Elsevieres. [Online]. [29 September 2016]. Available from: http://external.elsevier.es/espacioformacion/eimc/temas/m2t8.pdf
- Ediciones el paÃs. 1999. EL PAÃS. [Online]. [29 September 2016]. Available from: http://elpais.com/diario/1999/06/02/sociedad/928274421_850215.html
- Utahedu. 2016. Utahedu. [Online]. [29 September 2016]. Available from: http://learn.genetics.utah.edu/content/labs/microarray/
Página web de interés: Genomegov. 2016. National Human Genome Research Institute (NHGRI). [Online]. [8 October 2016]. Available from: https://www.genome.gov/10000533/dna-microarray-technology/
Autora: Marina Gómez (2º Bachillerato)- Clonación géncia
Se trata de una técnica que se aplica a los genes que componen el ADN, en la cual se selecciona un gen de interés de un organismo y se extrae para introducirlo en una bacteria que pueda replicarlo, un vez hecho se cultiva la bacteria para obtener un determinado número de bacterias que expresen ese gen.
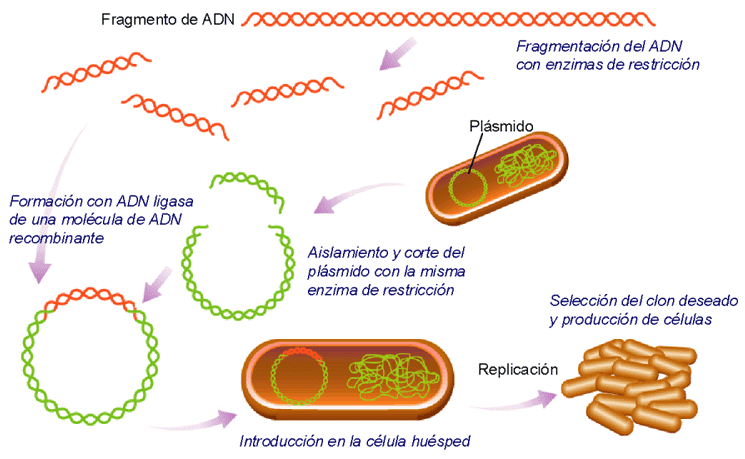
Para llevar a cabo exitosamente este proceso hay que seguir ciertas etapas:
- Preparación del ADN: En primer lugar se debe seleccionar el gen que se quiere implantar y se debe extraer rompiéndolo con un enzima de restricción.

- Preparación del vector: Para introducir el gen deseado en el interior de una bacteria es necesario un vector que lo transporte. Este puede ser un virus, un plásmido o un cósmido, al cual hay que retirar un fragmento de ADN con la misma enzima de restricción que rompió el ADN del donante del gen.
- Unión del ADN al vector: A continuación, Se une el gen deseado al vector, con la ayuda de un enzima ligasa.

- Incorporación del ADNr: Esta es la etapa más importante. En ella se introduce el vector en el interior de la bacteria, y al replicarse, también lo hace el material genético implantado.

- y 6. Propagación celular y selección: Estas dos etapas deben realizarse a la vez. Debe comprobarse qué bacterias han replicado el ADN recombinante, ya que no todas lo hacen y aislar a las que si lo han replicado para trasladarlas a una zona de cultivo. De este modo conseguiremos un cultivo de bacterias únicamente con el gen implantado.
7.Expresión del ADN clonado: Finalmente, se debe comprobar que las bacterias obtenidas expresen el ADN implantado correctamente, para verificar que el proceso ha sido exitoso.
Esta técnica tiene enormes utilidades, como por ejemplo fabricación de insulina. Se introduce un gen que expresa la fabricación de insulina en una bacteria, esta se reproduce y obtenemos un gran número bacterias capaces de producir insulina humana para las personas que sufren diabetes.
En resumen, su principal objetivo es el de conseguir que se exprese un gen masivamente con el fin de obtener lo que ese gen exprese sin necesidad de obtenerlo directamente de otros seres que no solo son perjudicados sino que además las cantidades que se extraen son mucho más pequeñas comparadas con las que se pueden obtener con este método.
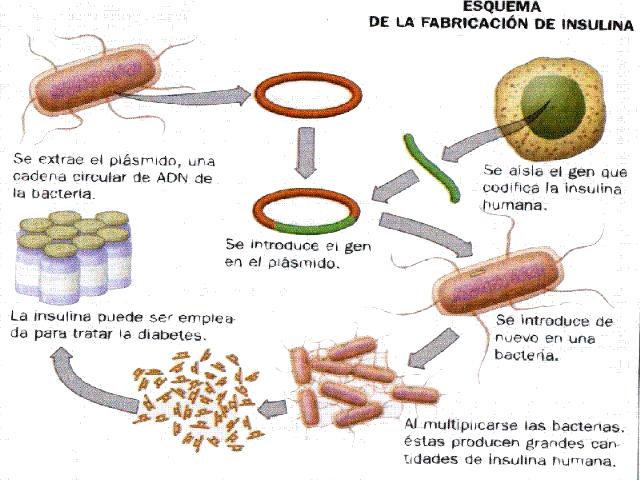
Bibliografía:
http://www.peybur.com/clonacion-genetica.htmlhttp://profedenaturales.blogspot.com.es/2012/06/experimento-o-proyecto-clasico-de.html
Vídeo que muestra el proceso de clonación génica:
Autor: Javier González (2º Bachillerato)- Cristalografía de rayos X
Cristalografía de rayos X
La cristalografía de rayos X es una técnica que nos permite ver estructuras a nivel atómico, es decir, es como una microscopía de muy alta resolución.La cristalografía de rayos X fue esencial en la descripción de la doble hélice de la molécula de ADN . Actualmente, uno de sus principales usos es para estudiar la estructura de las proteínas
Se basa en la difracción de los rayos X por cristales. Los rayos X son ondas electromagnéticas de una longitud de onda muy pequeña (0,1nmm). Estos interactúan con los electrones que rodean los átomos ya que tienen la misma longitud de onda. Son dispersados por las nubes electrónicas de los átomos que forman parte del cristal dando lugar a un determinado patrón de difracción (este patrón consiste en miles de puntos) que puede interpretarse según la ubicación de los átomos en el cristal, aplicando la ley de Bragg.

Existen tres métodos para producir difracción de rayos X.
- Método de Laue: los rayos X inciden en un cristal, esto hace que de cada longitud de onda, exista un determinado ángulo. Se coloca la película detrás del cristal. En este método, los haces difractados forman un patrón de machas circular o elíptico.

- Método de rotación de cristal: los rayos X monocromáticos inciden en el cristal. La película se envuelve, rodeando al cristal. El cristal se hace girar sobre el eje perpendicular al haz incidente, el cual coincide con el eje del cilindro. Para encontrar el ángulo al cual se cumple la ley de Bragg, el giro del cristal se hace sucesivamente de 0° a 90°, hasta encontrar el patrón de difracción.
.jpg)
- Método del polvo
El cristal se reduce a polvo de tal manera que se forma un conjunto de pequeños cristales. Al hacer girar este cristal de 0° a 360°, el haz forma un cono. El interés de este método es formar este cono con las distintas orientaciones posibles de los diferentes cristales.
Bibliografía:
- Fuente principal:
- Otras fuentes:
Autora: Lucía Saiz (2º de Bachillerato)- Cromatografía
¿Qué es la cromatografía?
La cromatografía es un método físico que se utiliza en los laboratorios para diferenciar los componentes de una mezcla, para poder diferenciarlos o para medir la cantidad de cada componente en la mezcla.
¿Cómo se hace?
El método se basa en el principio de selección selectiva, que consiste en depositar una muestra en una fase móvil que consiste en un fluido que reparte la mezcla por la fase estacionaria, que consiste en un sólido o líquido pegado a un sólido. Los componentes repartidos en la fase estacionaria por la fase móvil, interaccionara de distinta forma con esta, diferenciando sus componentes.
Tipos de cromatografías
Las cromatografías se clasifican de dos formas: dependiendo del proceso de separación que ocurre cuando interaccionan las dos fases o dependiendo de la manera de poner las fases en interaccionan.
Dependiendo del proceso de separación
-Cromatografía líquida: Tienen la fase móvil está en estado líquido y existen varios tipos dependiendo del estado de la fase estacionaria:
A) Con fase estacionaria sólida tenemos:
1-Cromatografía de adsorción: La fase estacionaria diferencia los solutos por adsorción física y química causada por interacción de fuerzas de van der Waals.
2-Cromatografía de cambio iónico: La fase estacionaria se carga electrostáticamente atrayendo por diferencia de cargas los iones de los solutos en la carga móvil. Aquí un vídeo de cromatografía iónica
3-Cromatografía de exclusión: Se coloca la mezcla sobre una fase estacionaria porosa por la que pasan algunas moléculas y otras no dependiendo de su tamaño. Aqui video explicativo
B) Con fase estacionaria líquida tenemos:
1-Cromatografía de reparto: Diferencia los solutos de la mezcla según su solubilidad en la fase estacionaria líquida
2-Cromatografía de afinidad: Diferenciación de algún componente de la mezcla por su tendencia adherirse al compuesto que se use en la fase estacionaria. Aqui video informativo
-Cromatografía de gases: La fase móvil está en estado gaseoso y existen las siguientes variaciones:
1) Cromatografía de adsorción: Se coloca una fase estacionaria sólida que retiene los componentes de la fase móvil gaseosa de la misma forma explicada anteriormente.
2) Cromatografía de reparto: Se vierte el gas en una fase estacionaria líquida que retiene algunos gases por impregnación
Dependiendo de la forma de interacción entre las fases
-Cromatografía en columna: Se coloca la fase estacionaria en un tubo estrecho por el que se hará pasar la fase móvil por presión, capilaridad o gravedad en un estado líquido o gaseoso.
-Cromatografía plana: Se coloca una placa fina como fase estacionaria por la que se verterá la fase móvil en estado líquido o gaseoso, diferenciando los solutos por capilaridad y gravedad.
Biliografía
Es.wikipedia.org. (2016). Cromatografía. [online] Available at: https://es.wikipedia.org/wiki/Cromatograf%C3%ADa [Accessed 23 Sep. 2016].
perfil, V. (2012). Cromatografía. [online] Quimicaterceroliceo42.blogspot.com.es. Available at: http://quimicaterceroliceo42.blogspot.com.es/2012/05/cromatografia.html [Accessed 23 Sep. 2016].
'Cromatografia Qué Es, Fases, Tipos Y Ejemplos Sencillos' (Areaciencias.com, 2016) <http://www.areaciencias.com/quimica/cromatografia.html> accessed 8 October 2016
perfil V, 'Cromatografía' (Quimicaterceroliceo42.blogspot.com.es, 2012) <http://quimicaterceroliceo42.blogspot.com.es/2012/05/cromatografia.html> accessed 8 October 2016
(2016) <http://www4.ujaen.es/~mjayora/docencia_archivos/Quimica%20analitica%20ambiental/Tema6.pdf> accessed 8 October 2016
'Cromatografía' (Biomodel.uah.es, 2016) <http://biomodel.uah.es/tecnicas/crom/inicio.htm> accessed 8 October 2016
Autor: Andrés Cano (2º Bachillerato)- D
- Drosophila melanogaster
La mosca de la fruta, o del vinagre, conocida como Drosophila melanogaster , es un pequeño insecto que pertenece al orden de los dípteros (el grupo de las moscas). Como la gran mayoría de las moscas, son inofensivos, aunque su costumbre cropófaga no les ha dado buena fama. Sin embargo, esto no ha impedido que la mosca del vinagre se haya hecho un lugar en el laboratorio y que, a estas alturas, sea uno de los organismos modelo de mayor renombre en la investigación.
HISTORIA
1901
William E. Castle realiza las primeras experiencias conDrosophila melanogaster en el laboratorio. Thomas H. Morgan y otros investigadores también empiezan a utilizarla los primeros años del siglo XX.
1910
El grupo de Thomas H. Morgan alcanza los primeros mutantes de caracteres concretos y definidos, el más conocido es white (color de ojos). La experimentación se decanta hacia la investigación genética.
1913
Alfred H. Sturtevant construye el primer mapa genético de un cromosoma de Drosophila melanogaster. Seguidamente, se construirán los otros tres.
1915
Thomas H. Morgan publica el libro "El mecanismo de la herencia mendeliana" donde se explica el papel de los cromosomas como material hereditario.
1927
Hermann Müller utiliza una técnica que utiliza rayos X para inducir mutaciones "a gran escala" en la mosca de la fruta. Muchos de los mutantes mantienen el fenotipo en las siguientes generaciones.
1933
Emile Heitz y H. Bauer describen los cromosomas politénicos de las glándulas salivales en la mosca de la fruta.
1940
Conrad Waddington determina los genes implicados en el desarrollo del ala de la mosca de la fruta.
1946
Edward B. Lewis propone que los genes bithorax que se expresan en un segmento dan identidad a un segmento que se encuentra a nivel posterior.
1969
Joseph G. Gallo y Mary-Lou Pardue realizan las primeras hibridaciones in situ de los cromosomas politénicos.
1974
David Hogness y Michael Grunstein crean la biblioteca genómica de la mosca de la fruta, es decir, el conjunto de secuencias representativas de su genoma.
1979
Edward B. Lewis obtiene moscas con dos segundos segmentos torácicos y dos pares de alas, en vez de uno. Es la primera clonación posicional del gen ultrabithorax.
1980
Christiane Nüsslein-Volhard y Eric Wieschaus realizan un análisis de mutantes en todo el genoma de la mosca de la fruta e identifican los genes implicados en el desarrollo del embrión.
1982
Gerald M. Rubin y Allan C. Spradling producen las primeras moscas transgénicas.
1983
El laboratorio de Walter Gehring, en Suiza, y el de Thom Kaufman, con Matthew Scott y Amy Weiner, en Estados Unidos, descubren de manera independiente la existencia de los genes Homeobox a la mosca de la fruta.
1993
Andrea Band y Norbert Perrimon desarrollan el sistema de dos componentes para controlar la expresión génica en la mosca de la fruta.
Autor: Guillermo Martín-Aragón2000
Secuenciación completa del genoma de la mosca de la fruta.

PRIMEROS EXPERIMENTOS
Los primeros usos de esta especie se centraron en estudios experimentales sobre evolución. Su corto ciclo vital (de 10 a 15 días) con la generación de numerosas familias de individuos permitía estudiar la aparición y transmisión de mutaciones en sucesivas generaciones sometidas a diferentes condiciones ambientales (temperatura, humedad). A partir de 1910, cuando el grupo de Morgan encontró los primeros mutantes de caracteres claramente definidos y heredables (color de ojos, forma de las alas, etc.), como los famosos mutanteswhite de ojos blancos, fue cuando se vio su idoneidad para llevar a cabo estudios genéticos.
GENOMA
El genoma de la mosca de la fruta fue completamente secuenciado en marzo del año 2000, gracias al consorcio público y la empresa Celera Genomics. Contiene 180 millones de pares de bases (Mb) que albergan alrededor de 13.6000 genes, aproximadamente un tercio de los que contiene el genoma humano. Su secuencia es de acceso público y los investigadores pueden buscar las correspondencias entre los genes de la mosca y los genes involucrados en ciertas enfermedades humanas. La era de la genómica, pues, ha abierto un nuevo contexto para la investigación sobre la mosca de la mosca que la vincula a la investigación biomédica.

CULTIVO DE LA MOSCA
https://www.youtube.com/watch?v=XoIrjGLR8Yc
BIBLIOGRAFIA
http://es.slideshare.net/5554382/mutaciones-morfolgicas-en-drosophila-melanogaster
http://www.biology-pages.info/D/Drosophila.html
http://www.botanical-online.com/animales/mosca_vinagre.htm
- E
- ELISA
Las pruebas ELISA (ensayo por inmunoabsorción ligado a enzimas) se valen de las reacciones antígeno-anticuerpo. Estas pruebas se utilizan en diversos campos, como son el de la medicina, la biología o la bioquímica. Las pruebas ELISA se utilizan por ejemplo para diagnosticar el VIH o la hepatitis B, para detectar gérmenes, antígenos en la orina o anticuerpos que estén atacando microorganismos.
¿Cómo se realizan estas pruebas?
En una serie de pocitos se colocan anticuerpos o antígenos. En estos pocitos se vierten las diversas sustancias en las que se quiere descubrir si existen determinados antígenos o determinados anticuerpos, si es así el antígeno se une al anticuerpo (o viceversa), entonces se añade una enzima que coloreará la muestra si la unión se ha producido, lo que querrá decir que el antígeno o el anticuerpo que buscábamos se encontraba en la muestra.El cambio de color de la muestra se mide con un espectrofotómetro.
Algunas de las enzimas más utilizadas como marcadoras son: peroxidasa, glucosa oxidasa y fosfasa, entre otras.

Tipos de ELISA : método sandwich, método de unión competitiva, método indirecto y método directo.
Bibliografía ELISA y métodos :
Silva, M.C; García, M.J. Técnico Especialista en Laboratorio del Servicio de Salud de la Comunidad de Madrid Temario Específico Volumen 2. Convocatoria 2015, 7 editores, Sevilla, 2015.
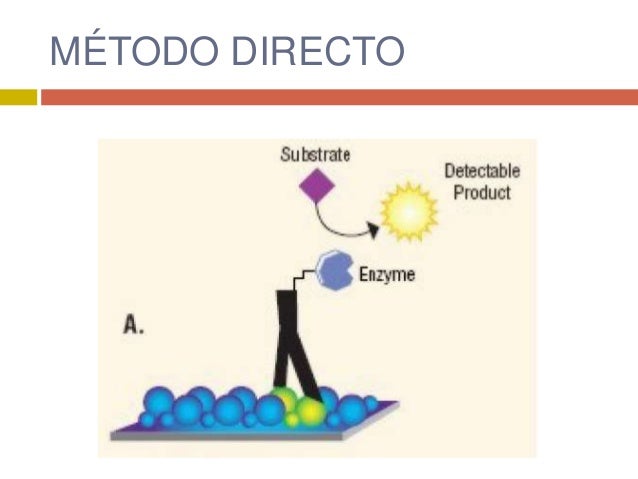
Método directo
Este método se utiliza para detectar antígenos. Sigue los siguientes pasos:
1- Se vierte la solución en la que se busca el antígeno en los pocillos.
2-Se lleva a cabo una incubación con anticuerpos específicos marcados con enzimas. Se marcará la muestra si esta es positiva.
Método indirecto
Se trata de un tipo de ELISA que se utiliza para detectar anticuerpos. Sigue los siguientes pasos:
- Se pone en el pocito el antígeno específico del anticuerpo que buscamos.
- Se añade la sustancia en la que buscamos el anticuerpo.
- Se añade una antiglobulina que ha sido marcada con una enzima.
- Se observa el cambio de color en la muestra.
El método sandwich es un tipo de ELISA, que sirve para detectar antígenos y anticuerpos.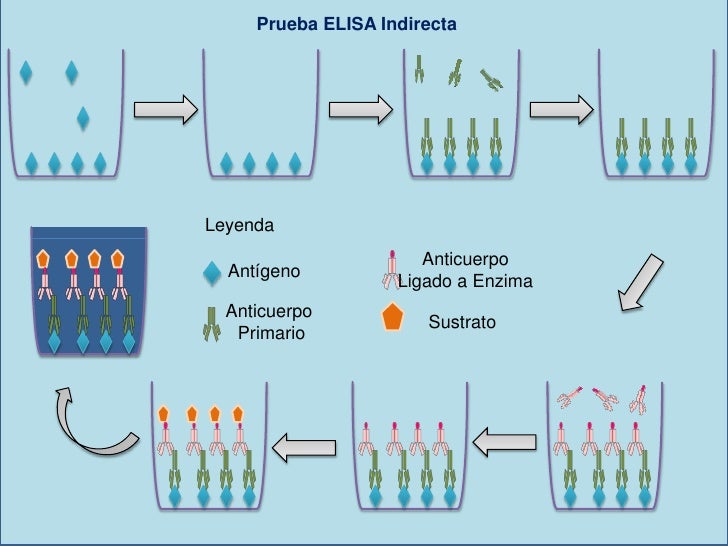
Para detectar antígenos: sirve para detectar antígenos de gran tamaño, (virus de la hepatitis, por ejemplo). Este método se divide en varias fases:
- Se pone en el soporte el anticuerpo específico para el antígeno que buscamos.
- Se añade la muestra que estamos estudiando.
- Se añade el anticuerpo específico para el antígeno que estamos buscando pero, esta vez, marcado con una enzima.
- Se añade una sustancia incolora, específica para la enzima marcadora, que cambiará de color si esta es positiva.
Para detectar anticuerpos el método sandwich seguirá los siguientes pasos:
- Se pone una antiglobulina del anticuerpo que buscamos en el soporte.
- Se añade la muestra en la que estamos buscando el anticuerpo.
- Se añade un antígeno específico para el anticuerpo.
- Se añade el antígeno que estamos buscando pero este marcado con una enzima.
- Estudiamos el cambio de color producido.
Método de unión competitiva
Se trata de un tipo de ELISA. Este método se utiliza para la detección de drogas y hormonas (principalmente) en fluidos biológicos.
Para detectar antígenos se siguen las siguientes pautas:
- Se pone el anticuerpo específico del antígeno que buscamos en el pocito.
- Se añade la mezcla que resulta de juntar la sustancia en la que buscamos el antígeno y una solución con ese mismo antígeno pero este marcado con una enzima.
- Se mide la cantidad del antígeno marcado que se ha unido a los anticuerpos del pocito. En este método la cantidad de antígeno que buscamos sera inversamente proporcional a la coloración de la mezcla.
Este método también se usa para detectar anticuerpos, en este caso se siguen los mismos pasos que para detectar antígenos.
Para saber más
Bibliografía ELISA y métodos :
Silva, M.C; García, M.J. Técnico Especialista en Laboratorio del Servicio de Salud de la Comunidad de Madrid Temario Específico Volumen 2. Convocatoria 2015, 7 editores, Sevilla, 2015.
Autora: Rocío Gómez (2º Bachillerato)- Electroforesis
- UTILIDAD:
¿Qué es ?, ¿para qué sirve?.
La electroforesis es un tipo de técnica de laboratorio que sirve para separar moléculas en una disolución cuando están sometidas a un campo eléctrico. Es decir, para poder entenderlo, en una placa con un polo cargado positivamente y el otro de forma negativa. Al colocar las muestras obtenidas, las moléculas con más cargas positivas se dispondrán de forma gradual en el polo negativo y las que estén cargadas de forma negativa en el polo positivo.
¿Cómo lo hace?
La muestra debe colocarse sobre un soporte. Actualmente, el más utilizado es el soporte de gel, formado por polímeros creando una especie de malla, tiene unos pequeños pozos, lugar donde se ponen las muestras para evitar que éstas se mezclen y las prueba sea fallida. Las moléculas principalmente se separan por factores de fuerza y tamaño.
Hay 3 tipos de electroforesis:
- De frente móvil: las muestras están por toda la disolución y se observa sus colocaciones frente a ésta.
- Zonal: las muestras se aplican en los pozos en forma de bandas verticales y a través del gel las muestras se separan simplemente.
- Continua: la muestra se aplica en toda la base pero de forma continua, durante todo el proceso.

En el siguiente vídeo se aporta una prueba de electroforesis en gel de agarosa. Esta prueba se hizo parecida el curso 2016/17 en nuestro mismo instituto.
Como sitio web de interés aquí os dejo esta pagina para poder profundizar más en esta técnica de laboratorio.
Autora: Celia Carrero (2º Bachillerato)- Escherichia coli
-
La Escherichia coli, es una enterobacteria, que habita en los intestinos de los animales y las personas. Su nombre se debe a su descubridor, Theodore von Escherich.
Es la encargada de fermentar la lactosa y la glucosa con producción de ácidos y gases. Su temperatura óptima es de 37ºC, aunque puede crecer a 45ºC, lo que lo diferencia de otras bacterias coliformes. Crea colonias de un tamaño de 2-3mm y de aspecto variable. Las colonias típicas son lisas, poco elevadas, grises, de borde entero, y con la superficie brillante. En cambio las colonias rugosas son secas y difíciles de dispersar. Esta bacteria, vive en simbiosis con el hombre y le es beneficioso, puesto que sintetiza vitamina K y parte del complejo vitamínico B. En el intestino humano de un mismo individuo no existen todos los tipos de colibacilos, solamente 3 o 4, de los cuales 1 o 2 son habituales y los otros se consideran pasajeros. El cambio de una raza residente en el intestino humano suele ir acompañada de un estado diarréico.
Entre las infecciones oportunistas extraintestinales ocasionadas por el E.coli destacan:
-Infecciones urinarias (cistitis, pielonefritis).
-Infecciones biliares (colescistitis), peritoneales y meningitis.
-Infecciones de las heridas (abscesos) y mucosas (otitis, sinusitis).
-Septicemias.




- F
- G
- H
- HPLC (Cromatografía Líquida de Alta Precisión)
HPCL (Cromatografía líquida de alta precisión)

Para hablar de cromatografía de alta precisión primero debemos saber qué es la cromatografía:
Y es un grupo de técnicas utilizadas para separar sustancias por medio del desplazamiento selectivo y a diferentes velocidades. Sirve para separar, identificar y cuantificar los componentes orgánicos e inorgánicos de una muestra.
La HPCL suele separar gases con una volatilidad muy baja y con pesos moleculares bastante grandes. Se emplean dos fases en esta técnica, una móvil y otra estacionaria.
Es una técnica sencilla, rápida y precisa y con útil para analizar una gran variedad de sustancias.
Los recipientes de la fase móvil suelen ser de vidrio o de acero inoxidable, y contienen el disolvente.
Las bombas que se suelen utilizar pueden ser de tres tipos:
-Jeringa remachada con tuerca
-Bombas de vaivén
-Neumáticas o de presión constante
La máquina también posee diversos detectores para evaluar las sustancias.
En la fase móvil, los componentes de la muestra se separan. Después se disuelven en el disolvente y a continuación pasan por las columnas mediante la presión ejercida por el bombeo. Posteriormente, en la fase estacionaria, cada uno sale en momentos distintos.
Esta técnica se emplea en numerosos campos, como biotecnología, ciencias y la industria farmacéutica.
BIBLIOGRAFÍA
https://prezi.com/vqqy_kalnusj/cromatografia-liquida-de-alta-resolucion-hplc/
Autor: Julio Raúl Cordal (2º Bachillerato)- Hibridomas
- Nuestro cuerpo no siempre produce anticuerpos para combatir agresiones tanto externas, como internas (cáncer). Por suerte, en 1975 un médico argentino, Cesar Milstein, creó el primer Hibridoma,(1) pero esta creación no se evidenció hasta 1987 cuando éstos fueron usados en ratones; más tarde fueron usados en diagnósticos.(2)Un hibridoma se obtiene de la fusión de células plasmáticas tumorales con un linfocito B sano (productor de anticuerpos), dando lugar a grandes cantidades de anticuerpos monoclonales, es decir, que se unen a un antígeno específico.(3)
Los anticuerpos monoclonales se utilizan para diversos procedimientos como la identificación de antígenos tumorales y antígenos infecciosos, la medición de niveles de proteínas y drogas presentes en el suero, cuantificación de hormonas... (2)
¿Cómo se obtienen estos hibridomas?

Bibliografía
- Texto
http://elpais.com/diario/1980/12/19/sociedad/346028409_850215.html
http://www.quimicaviva.qb.fcen.uba.ar/contratapa/aprendiendo/capitulo10.htm
Autora: Laura Rodríguez (2º de Bachillerato)
- I
- J
- K
- Knout génico
Técnica de laboratorio usada para bloquear un gen en concreto para que no se exprese en un organismo, y así descubrir su función. Esto se consigue sustituyéndolo por una versión del mismo al que se le quitan uno o varios axones y así conseguir que sea incapaz de producir la proteína que normalmente si produciría.1
El método a seguir empieza preparando un plásmido que contenga el gen de interés. Después se introduce en una célula embrionaria de por ejemplo un ratón. Ya dentro de la célula y después de la recombinación genética el gen se cambia por el de interés. Las células de este ratón se introducen en el útero de un ratón hembra. Y finalmente dará lugar a un ratón knock out con el inactivo. 2

vídeo en inglés sobre knock out
bibliografía:
Autor: Diego Soto (2º Bachillerato)
- L
- Levadura
El término levadura proviene del latín "levare", que significa levantar, ya que produce dióxido de carbono que expande las proteínas del gluten en la harina.
Las levaduras son hongos unicelulares pertenecientes al Reino de los Hongos.
Forman colonias de células dotadas de una pared gruesa.
Se reproducen asexualmente por gemación. Su tiempo de reproducción varía entre especies, pero está entre las 2 o 3 horas en condiciones normales de crecimiento.

HÁBITAT:
Las levaduras con ubicuas, se encuentran en suelos y materia vegetal en descomposición.
Aunque necesitan condiciones anaerobicas para fermentar, es posible verlas crecer en medios de cultivo líquido y sólido en una proporción atmosférica de oxígeno de un 21%.
En el medio sólido no utilizan la fermentación como via catabólica, sino que respiran y usan el ciclo de Krebs.
APLICACIONES:
Además de producir proteínas, estan contienen alta cantidad de aminoácidos, péptidos, carbohidratos y sales; que al extraerse por medio de una disolución acuosa se crea un extracto de levaduras. Este extracto cuya propiedad es antioxidante, es utilizado como saborizantes en sopas, salsas, condimentos y como suplemento alimenticio en la comida de algunos animales(cerdos, aves).
Son los organismos más modificados genéticamente, aunque nunca se llevan a cabo, ya que no estan autorizadas para el consumo humano.
ESPECIES:
Conocemos alrededor de 500 especies de levaduras.
Fueron descubiertas por primera vez por su capacidad de fermentación por Louis Pasteur en 1857.
SACCHAROMYCES CEREVISIAE: Especie más conocida y utilizada, que significa "levaura comedora de azúcar". Esta levadura lleva a cabo la fermentación alcohólica, de la que se obtienen productos para el consumo humano, como el vino, el vinagre, la cerveza o el pan. Es el primer genoma eucariótico secundario.

BIBLIOGRAFÍA:
Libro de Biologia de 2ºBachillerato SM
Biología.laguia2000.com
Sabermaz.umich.mx
Autora: Jessica Susé (2º Bachillerato)
- M
- Microscopio confocal
¿Para qué sirve?
La microscopía confocal es pareciada a la microscopía epifluorescencia, pero más desarrollada, ya que permite analizar toda la información de la muestra de manera tridimensional, tanto si el tejido de esta es grueso o más fino.1 Lo más llamativo de esta microscopía es que permite capturar imágenes con una elevada calidad gracias al desarrollo de la microscopía de fluorescencia, pues al obtener técnicas mucho más avanzadas de filtración, estas técnicas absorben la luz de alrededor de los focos y la eliminan, proporcionando así que el microscopio se centre en un punto de la muestra teniendo una gran calidad, pues antes no se apreciaban bien las muestra porque estaban "sucias" por la luz de los focos. 2
La evolución de la microscopía y el descubrimiento de la proteína fluorescente GFP, permite estudiar las funciones y las estructuras de las proteínas en las células vivas, y ver los tejidos microscópicos en vivo.
Esta microscopía tiene muchas aplicaciones: en biología molecular, biología vegetal, medicina, fisiología, inmunología... 3
¿Cómo se hace?
El microscopio confocal es un aparato que tiene un foco de luz, que suele ser un láser. El láser emitido atraviesa un espejo dicróico y una lente objetivo hasta llegar a la muestra, al llegar se estimula la fluorescencia de la misma haciendo que esa fluorescencia sea "agrupada" por la lente citada anteriormente y reflejada por el espejo dicroico que la conduce al detector, el cual no deja pasar la fluorescencia, solo la luz que procede del plano. 1
Bibliografía:
1. Microscopía
3. Satellite
Para más información:
Autora: Ana María Benítez (2º Bachillerato)- Microscopio electrónico de barrido.
¿Qué es y para qué sirve?
La microscopía electrónica de barrido o SEM, se basa en el principio de la microscopía óptica en la que se sustituye el haz de luz por un haz de electrones. Fue inventada por Manfred von Ardenne en 1937. Con esta técnica se consigue una resolución muy superior a cualquier otro instrumento óptico, llega hasta los 100 Angstrom aproximadamente.

Microscopio electrónico de barrido. Fuente: http://casanchi.com/ref/mbe01.htm
¿Cómo lo hace?
Su funcionamiento consiste en un instrumento capaz de ofrecer un variado rango de informaciones procedentes de la superficie de la muestra. Se basa en barrer un haz de electrones sobre un área del tamaño que deseemos mientras en un monitor se visualiza la información que hayamos seleccionado en función de los detectores que hayan disponibles. Esta técnica es muy útil ya que pueden ampliar los objetos 200.000 veces y además produce imágenes tridimensionales de la superficie del objeto.

Partes del microscopio. Fuente: http://b.se-todo.com/biolog/16666/index.html
Tipos de detectores
- Detector de electrones secundarios (SE)
- Detector de electrones retrodispersados (BSE)
- Detector de rayos X (EDS)
- Detector de rayos X (WDS)
- Detector de electrones retrodispersados difractados (BSED)
¿Qué aplicaciones tiene en los campos?
Gracias a sus análisis podemos obtener datos como textura, tamaño y forma de la muestra. Entre las áreas de aplicación de esta técnica, se pueden mencionar: geología, estudio de materiales, metalurgia, odontología, arqueología, fibras textiles, medicina, medicina forense, botánica, biomedicina, peritaciones caligráficas, química y electrónica.
- Si quieres obtener más información sobre la microscopía electrónica de barrido puedes acceder a esta página web: http://www2.uned.es/cristamine/mineral/metodos/sem.htm
- Vídeo: https://youtu.be/P7yOTrd0l2M
Bibliografía:
http://www.upv.es/entidades/SME/info/753120normalc.html
https://www.ecured.cu/Microscopio_electr%C3%B3nico_de_barrido
Autora: Lyia Mihailova (2º Bachillerato)- Microscopio electrónico de transmisión
¿Para que sirve?
El microscopio electrónico de transmisión (MET) permite el estudio a nivel ultra estructural de material biológico (células y tejidos tanto animales como vegetales). Permite también la caracterización morfológica de materiales de forma directa, así como su composición mediante técnicas espectroscópicas asociadas. Aporta además información funcional sobre el material sujeto a estudio.
Imagen 1. Estructura del microscopio electrónico de transmisión .
¿Cómo lo hace?
Utiliza un fino haz de electrones acelerados a gran velocidad como fuente de iluminación. Dichos electrones atraviesan la muestra, produciéndose la dispersión de los mismos en diferentes trayectorias características de la ultra estructura del material observado.
La imagen de alta resolución formada es aumentada y proyectada sobre una pantalla fluorescente para su visualización en tiempo real, pudiendo registrarse digitalmente o en negativos para su estudio posterior.
La estructura de un microscopio electrónico de transmisión esta indicada en la Imagen 1, indicando cada uno de sus componentes y el trayecto que recorre el haz de electrones para llegar a la muestra proporcionar su visualización.
Imagen 2: Una microscopía electrónica del poliovirus.
En la Imagen 2, se puede visualizar el resultado de una muestra visualizada mediante el microscopio electrónico de transmisión, como se puede observar, en blanco y negro, indicando la estructura de dicha muestra, en este caso, del poliovirus.
A continuación, se indicaran sus diversas aplicaciones:
1.- Ciencia de materiales.
2.- Biología Celular.
3.- Biomedicina
4.- Microbiología.
5.- Biología Vegetal y Animal.
Realmente, esta herramienta nos ha permitido y nos sigue permitiendo, descubrir estructuras del mundo celular, donde podemos corregir algunas malformaciones y ajustarlas a nuestro gusto o, corregirlo como es debido.
Si quieres ver de manera más detallada y visual la información de esta herramienta, te recomiendo que visites este vídeo de la Universidad de Navarra, pinchando sobre el siguiente título:
Procesamiento de muestras para Microscopía ElectrónicaBibliografía:
Autor: Petyo Andrianov Peychev- Mosca del vinagre (Drosophila melanogaster)
-
La Drosophila melanogaster, es una mosca conocida más comúnmente como la mosca del vinagre o la mosca de la fruta, recibiendo este nombre debido a que se alimenta de fruta fermentada. Pertenece a la familia Drosophilidae.
.jpg/270px-Drosophila_melanogaster_-_side_(aka).jpg)
Normalmente este tipo de insecto se usa para investigaciones genéticas ya que posee un número reducido de genomas cromosomas (4 pares), su ciclo de vida es reducidamente corto (entre los 15 y los 21 días), además, un alto porcentaje de los genes de enfermedades (conocidas) que sufren los humanos se ven identificados con el genoma de esta mosca y muchas secuencias proteínicas de la Drosophila melanogaster se encuentran muy similares en los mamíferos. Fue adoptada como animal de investigación genética por Thomas Morgan (siglo XX).
Desarrollo de la Drosophila: https://www.youtube.com/watch?v=gzmyIsdHdA0
Más información:
http://www.seresmodelicos.csic.es/mosca.html
http://www.botanical-online.com/animales/mosca_vinagre.htm
http://www.univalle.edu.co/~geteg/5.html
Laura Rico Serrano 2ºBC.
- N
- Ñ
- O
- Optogenética
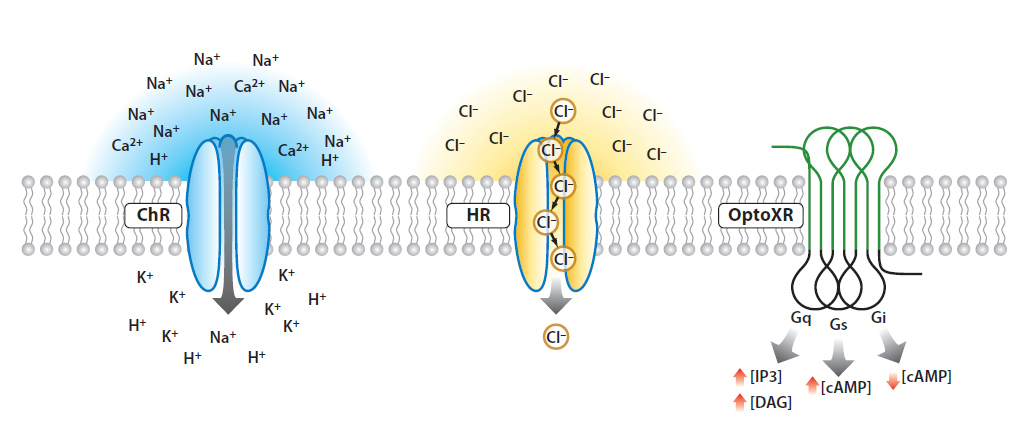
¿Qué es ?
Es un técnica que combina métodos ópticos y genéticos de tal manera que es posible controlar células y tejidos, dicho control es posible gracias a unas algas unicelulares sensibles a la luz que codifican proteínas fotosensibles de tal manera que procesan una respuesta en milésimas de segundos. Esta técnica está causando gran furor en enfermedades neurodegenerativas como el alzhéimer, autismo o depresión. (1)
Funcionamiento
La optogenética ,descubierta por el bioingeniero y psiquiatra Karl Deisseroth ,consiste en inyectar en grupos de neuronas virus benignos con proteínas fotosensibles .Pero esto no hubiera sido posible sin el descubrimiento de estas proteínas fotoreceptoras, llamadas opsinas, las cuales se han obtienen de un organismo unicelular ,el alga Chalamydomonas reinhardtii o también denominada Channelrodopsina2 (ChR2) la cuál tienen una capacidad de desplazarse hacía la luz.(2)
Este desplazamiento es posible debido a una estimulación de luz permitiendo que el ChR2 se abra generando un gradiente electroquímico en la célula debido al flujo de iones. De esta manera la células neuronales se vuelve sensible a la luz y cada vez que reciban un haz de luz o impulsos luminosos, las células responderán desplazándose ,estimulando o suprimiendo recuerdos o como tratamiento contra ciertas patologías.(3)
Procesos
En la optogenética el tipo de de moléculas fotosensibles que se utilizan se obtienen de bacterias generando canales iónicos de calcio, sodio o potasio .Cuando las opsinas reciben un haz de luz se abre un poro en la membrana de la célula permitiendo así el flujo de iones. De esta manera podemos hacer que las neuronas se activen o inhiban dependiendo el tipo del canal iónico .(4)
Tipos de canales iónicos
Más tarde se secuencia el gen que posee información codificada por bacterias para transferirlo a las neuronas. Este paso consiste en introducir virus en el cerebro de ratones los cuales transportan la información genética para producir una proteína y permitir la expresión de neuronas seleccionadas.(5)
Pero para poder activar estas proteínas debemos insertar una fibra óptica y electrodo para poder manipular la actividad en el cerebro. Una vez instalada la fibra utilizamos una estimulación por una onda de luz específica desde un láser ,de tal manera que cuando encendamos un interruptor o lo apaguemos ,las proteínas fotoreceptoras activaran las neuronas y el ratón presentará diversos comportamientos.(5)
Aplicaciones (6)
-Controlar ataques epilépticos.
-Manipulación en ciertas neuronas para evitar la adicción a las drogas.
-Regulación del sueño,lo que permite la investigación de fármacos para tratar la narcolepsia.
-Manipulación de latidos del corazón mediante impulsos de luz para bombear la sangre y evitar posibles infartos.
-Tratamiento contra el Parkinson
Video:
Funcionamiento de las Opsinas y su aplicación en la optogenética.
Bibliografía
http://www.investigacionyciencia.es/
https://biotecnologia.fundaciontelefonica.com
https://www.scientificamerican.com
Autora: Tamara Sánchez-Arquero (2º Bachillerato)
- P
- PCR (Reacción en cadena de la polimerasa)
- La reacción en cadena de la polimerasa (abreviado PCR por sus siglas en ingles: Polymerase Chain Reaction) es una técnica de laboratorio que consiste en obtener una cantidad ilimitada de copias de fragmentos de ADN partiendo de una sola copia. Fue desarrollada por Kary Mullis en 1986 y actualmente es una técnica muy usada para la amplificación de fragmentos de ADN lo cual permite identificar virus y bacterias e investigar sobre el ADN.Esta reacción es muy sencilla y solo requiere de un tubo de ensayo, un fragmento de ADN, la ADN-polimerasa, una cadena de nucleótidos que actúen como cebadores y calor.
 Las copias se realizan automáticamente en una maquina como la de la fotografía y se necesitan 2 horas para conseguir una cantidad útil de copias. La maquina es capaz de generar 100.000 millones de moléculas idénticas en una tarde.
Las copias se realizan automáticamente en una maquina como la de la fotografía y se necesitan 2 horas para conseguir una cantidad útil de copias. La maquina es capaz de generar 100.000 millones de moléculas idénticas en una tarde. Vídeo explicativo de todo el proceso
Vídeo explicativo de todo el proceso
Para saber mas pincha AQUI - Pez cebra
El pez cebra es científicamente conocido como Danio rerio, es un organismo modelo, es decir, una especie que ha sido muy estudiada para investigar distintos aspectos de la biología.
Físicamente, es un pez alargado y pequeño (3,5 cm de longitud) con un tono amarillento, atravesado horizontalmente por una serie de líneas azules oscuras presentes en todo el cuerpo menos en la zona ventral, donde el tono se aclara. Las hembras son más grandes que los machos, pero estos son de un color más brillante (tonalidad dorada).

Es un pez de agua dulce que habita en zonas no muy profundas, como estanques, ríos, lagos... y también suele aparecer en plantaciones de arroz, canales...
Su reproducción es ovípara, la hembra pone hasta 200 huevos en forma continua durante todo el año.
Como ya se ha dicho antes es un organismo modelo, pero no solo eso, sino que es uno de los mejores modelos biológicos para el estudio de nuevos fármacos (principalmente sobre alimentación). El pez cebra tiene una gran similitud genética con vertebrados más grandes entre ellos los humanos), además es fácil de manejar y son abundantes y rápidos al reproducirse. Al ser transparente es más fácil manipularlo, ya que se observa el efecto que los fármacos producen, evitando otras pruebas que se deberían de hacer con otros organismos como los ratones. También es más fácil de mantener y criar que otros organismos modelos como la mosca del vinagre y el gusano.

Recientemente unos estudios han validado a este pez como modelo animal válido en las enfermedades genéticas humanas.
Que curioso que un ser tan diminuto pueda darnos tantas respuestas y soluciones sobre nosotros mismos.
BIBLIOGRAFÍA:
Autor: Jorge Marcos (2º Bachillerato)- Proteína verde fluorescente (GFP)

Aequorea victoria - https://es.pinterest.com/pin/8796161742508963/
La proteína verde fluorescente o GFP (Green Fluorescent Protein), fue descubierta en 1996 por Osamu Shimoura con sus estudios sobre la medusa Aequorea victoria, que forma naturalmente proteínas con luminosidad. Esta característica luz verde se forma porque la GFP de la medusa convierte las señales luminiscentes de otra proteína, la aecuorina.
El biólogo estadounidense consiguió aislar del celentéreo por vez primera la proteína que emitía una luz en la zona verde del espectro visible (aecuorina). Gracias a este descubrimiento, actualmente se utiliza en biología molecular como marcador.

https://juancarlosvivancos.com/
Está constituida por 238 aminoácidos, que forman once cadenas beta, cuyo conjunto forma un cilindro, en el centro del cual se encuentra una hélice alfa.
En esta hélice hay tres aminoácidos consecutivos que forman un cromóforo natural, de forma que cuando la GFP es iluminada con luz ultravioleta, produce una brillante fluorescencia verde.

https://biotechspain.com/es/index.cfm
Martin Chalfie consiguió clonar e introducir en 1988 esta proteína en un gusano antes transparente para la biología. Así, este gen comenzó a introducirse en los laboratorios masivamente como marcador. El gran descubrimiento era que ahora se podía introducir y mantener espontáneamente en las células, órganos, organismos… (siempre que estén vivos), sin tener que añadir otros agentes o cromóforos, y visualizarlos con mucha precisión. Su código genético puede fusionarse a otras proteínas, proporcionando a éstas una fluorescencia extra, que sirve para poder seguir su actividad en directo, seleccionar y aislar aquellas células que producen la proteína. Algo antes impensable.
Más tarde, Roger Tsien desarrolló variantes de la proteína, produciendo moléculas que emitían luz a distintas longitudes de onda, ampliando así la gama de colores de las GFP.
Estos descubrimientos hicieron que Shimomura, Chalfie y Tsien fueran galardonados conjuntamente con el premio Nobel de Química de 2008.
Para saber más:
- http://www.scielo.org.ar/scielo.php?script=sci_arttext&pid=S0025-76802009000400015
- http://www.redalyc.org/articulo.oa?id=179214945007
Vídeos para aprender más sobre las GFP:
Bibliografía:
Autora: Begoña Adán (2º Bachillerato)
- Q
- R
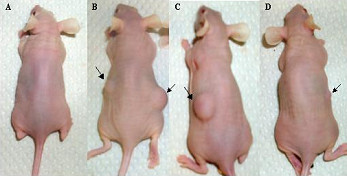
- Ratones desnudos
El ratón desnudo, también conocido como ratón atímico, es una de las especies animales mamíferas más utilizadas en el ámbito científico debido a su facilidad de uso por el tamaño, el conocimiento de todo su genoma, la fácil reproducción y por tener un sistema inmunitario deficiente. Al tener este tipo de sistema inmunitario significa que el ratón desnudo se trata de una mutación del ratón normal (Mus musculus) el cual posee más de 4000 genes. La mutación realizada a estos ratones se denomina nude (nu) causando la falta de pelo; y también el escaso número de lifoncitos T por la carencia de un timo funcional de estos animales.
Al carecer de lifoncitos T y tener un número de lifoncitos B normal, estos ratones sirven de gran ayuda para estudios sobre la actuación del timo en la diferenciación lifoncitaria; e investigaciones sobre las funciones e interacciones de los lifoncitos B con otras células inmunitarias como los macrófagos o Natural Killer.
Este tipo de ratones habitualmente se utilizan para las investigaciones sobre el cáncer y tratamiento de tumores; también sirven para la investigación sobre el sistema inmunitario y de enfermedades como el SIDA y la leucemia entre otras. Esto es porque al tener un sistema inmunitario tan deficiente, el ratón desnudo es capaz de recibir injertos mortales sin recibir rechazo al no tener la posibilidad de crear anticuerpos, ni poder desarrollar reacciones de hipersensibilidad.
Tan débil es su sistema inmunitario que incluso estos ratones pueden aceptar tejido injertado de otros organismos; como ocurrió en 1995 en el Centro Médico de la Universidad de Massachusetts, donde dos científicos, el Dr. Charles Vacanti y la Dra. Linda Griffith, fueron capaces de injertar un tejido de cartílago de oreja humano en un ratón desnudo haciéndole crecer una oreja en el lomo de este.
Esta mutación de ratones tenía muy poca esperanza de vida debido a su deficiente sistema inmunitario, impidiendo el avance científico relacionado sobre todo con el cáncer. No fue hasta la década de los setenta cuando otros dos científicos, John Stehlin y Beppino Giovanella, emplearon técnicas estériles logrando mantener vivo a uno de estos ratones desnudos durante dos años.
BIBLIOGRAFÍA.
Animalresearchinfo. 2016. Animalresearchinfo. [Online]. [30 november 2016]. Available from : http://www.animalresearch.info/es/el-diseno-de-la-investigacion/animales-de-investigacion/mouse-immunodeficient/
Fcenubaar. 2016. Fcenubaar. [Online]. [30 november 2016]. Available from : http://www.quimicaviva.qb.fcen.uba.ar/v5n1/carbone.pdf
Crivercom. 2016. Crivercom. [Online]. [30 November 2016]. Available from : http://www.criver.com/products-services/basic-research/find-a-model/athymic-nude-mouse
Envigocom. 2016. Envigocom. [Online]. [30 November 2016]. Available from : http://www.envigo.com/resources/data-sheets/envigo-5193-131-leaflets-2016-nude-mouse-combination-a4-refs.pdf
MÁS INFORMACIÓN SOBRE EL CASO DE GENERACIÓN DE UNA OREJA EN EL LOMO DE UN RATÓN DESNUDO: Blogspotcomes. 2016. Blogspotcomes. [Online]. [30 november 2016]. Available from : http://caballerosmolinienses.blogspot.com.es/2007/08/oreja-humana-en-el-lomo-de-un-raton.html
Autora; Cheyenne Montes (2º Bachillerato)
- S
- Secuenciadores
-
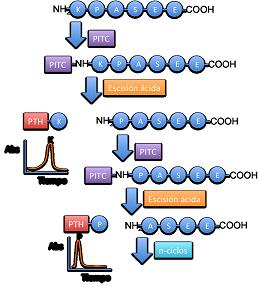
Un secuenciador es un aparato que sirve para determinar de forma automática la secuencia de los monómeros que componen un polímero lineal. Existen secuenciadores automáticos de ADN y de proteínas, que han disminuido la intervención humana en los procesos.
Secuenciar una proteína consiste en determinar la secuencia de aminoácidos que forman una cadena proteica. Mediante los secuenciadores se ha logrado automatizar este proceso, basado en el método de la Degradación de Edman. Al conocer la secuencia de aminoácidos de una proteína, podemos conocer las bases que codifican esa proteína, es decir, la estructura del gen correspondiente.

Secuenciar el DNA consiste en establecer el orden de nucleótidos de cualquier molécula. Para que el secuenciador de DNA comience a funcionar, necesita reconocer las cuatro bases de nucleótidos que componen el DNA: adenina, guanina, timina y citosina. Para ello, los científicos copian un trozo de DNA y añaden una pequeña cantidad de base fluorescente para cada lote de DNA. Cada base se unirá a su complemento de base en el extremo de una hebra. Los secuenciadores automáticos, en cambio, inyectan los lotes de DNA automáticamente en la parte superior del gel, aplican una carga negativa a un extremo del tanque, haciendo que las hebras migren a diferentes distancias, lo que marcará los distintos tamaños de hebras de DNA (electroforesis). Finalmente se necesita una fase de acabado para ordenar las secuencias obtenidas.


Para más información:
http://www.creces.cl/new/index.asp?imat=%20%20%3E%20%2014&tc=3&nc=5&art=653
Bibliografía:
http://www.biorom.uma.es/contenido/Glosario/S.html
http://www.ehowenespanol.com/funciona-secuenciador-automatico-adn-info_280237/
María Montero Lázaro
- Secuenciadores de ADN
La secuenciación ADN es el conjunto de reacciones bioquímicas que se producen en el ADN y que sirve para averiguar la secuencia de nucleótidos: adenina,timina, citosina y guanina (A T C G ) . Los métodos más utilizados y los más importantes son: la secuenciación automática y el método enzimático de terminación de cadena.
MÉTODOS CLASICOS DE SECUENCIACIÓN
Marcado: las moléculas que se quieren secuenciar son marcadas fluorescentemente o radiactivamente.
Separación: la cadenas de ADN marcada se diferencian en tamaño por una sola base. Estas cadenas de diferentes tamaños y longitudes se separan entre sí mediante electroforesis una técnica la cual separa moléculas según su movilidad en un campo eléctrico
Estas dos técnicas se consiguen mediante dos métodos : el método químico de Maxam que consiste en la rotura de cadenas sencillas de ADN marcadas radiactivamente , y el método enzimático de Sanger que consiste en el uso de un cebador el cual va a extender la cadena copiando de manera complementaria a la cadena molde de ADN .

SECUENCIACIÓN AUTOMÁTICA EMPLEANDO EL MÉTODO ENZIMÁTICO
Es un método similar al método de Sanger. Consiste en marcar el un cebador o los terminadores con un compuesto fluorescente y activar la reacción de secuenciación.

SECUENCIADORES AUTOMÁTICOS
Secuenciadores Automáticos Capilares: es una alternativa a los secuenciadores en geles desnaturalizados , consiste en un proceso muy similar a los geles pero en este se utiliza un polímero que se introduce o inyecta de forma automática en un capilar antes de cargar la muestra de la secuenciación.

Secuenciadores Automáticos mediante geles desnaturalizados
La secuenciación automática con geles desnaturalizantes, se realiza polimerizando un gel acrilamida en dos cristales montados .
APLICACIONES DE LOS SECUENCIADORES DE ADN
-Diagnostico enfermedades genéticas
-Desarrollo genoma humano
-Secuenciación ADN fósiles
BIOGRAFÍA E IMAGENES
http://www2.iib.uam.es/seq/tecnicas/biomed1.htm#metodos
http://www.cultek.com/img/otros/Aplicaciones/NGS/NGS003.jpg
http://pendientedemigracion.ucm.es/info/genetica/AVG/practicas/secuencia/Automat1.jpg
http://www.cultek.com/img/otros/Aplicaciones/NGS/NGS001.jpg
http://www.ehowenespanol.com/funciona-secuenciador-automatico-adn-info_280237/
LINK VIDEO YOUTUBE
<iframe width="300" height="169" src="https://www.youtube.com/embed/bSdHvT5lDs8?controls=0" frameborder="0" allowfullscreen></iframe>
Autor: Darío Lanzadera (2º Bachillerato)
- T
- U
- Ultracentrifugación
- La ultracentrifugación es una técnica utilizada para separar los componentes de una solución según su densidad y peso. Consiste en someter a las macromoléculas a un campo gravitatorio elevado tal que la fuerza de centrifugación sea mayor que la difusión consiguiendo así que se dispongan en capas (dependiendo de su coeficiente de sedimentación). Esta técnica es muy útil para averiguar el grado de homogeneidad o heterogeneidad de una solución al igual de la cantidad y el tipo de macromoléculas que se encuentran en la solución.La centrifugación puede darse en una escala preparativa o en una escala analítica. La analítica nos proporciona información acerca de las propiedades físicas (peso molecular o velocidad de sedimentación) y la preparativa nos permite separar y aprovechar las partículas según su velocidad de sedimentación (centrifugación diferencial), según su masa (centrifugación zonal) o su densidad ( centrifugación isopínica). En el primer caso obtendremos un líquido en la parte superior y materiales sedimentados en el fondo, en los otros caso las macromoléculas aparecerán distribuidas en capas.En la centrifugación de nucleótidos , por ejemplo, se utilizan dos tipos diferentes de centrifugación que son la centrifugación por velocidad de sedimentación y la centrifugación por equilibro(isopicnica).
- V
- W
- Western/Shoutern Blot
-
Southern blot, hibridación Southern o, simplemente, Southern es un método de biología molecular que permite detectar la presencia de una secuencia de ADN en una mezcla compleja de este ácido nucleico. Para ello, emplea la técnica de electroforesis en gel de agarosa con el fin de separar los fragmentos de ADN de acuerdo a su longitud y, después, una transferencia a una membrana en la cual se efectúa la hibridación de la sonda. Su nombre procede del apellido de su inventor, un biólogo inglés llamado Edwin Southern.
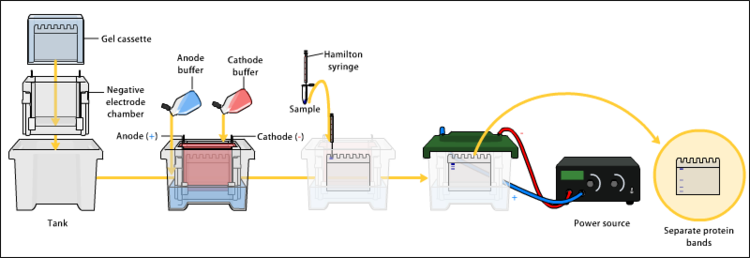
El Western blot, o inmunoblot, es una técnica analítica usada para detectar proteínas específicas en una muestra determinada. Mediante una electroforesis en gel se separan las proteínas atendiendo al criterio que se desee: peso molecular, estructura, hidrofobicidad, etc. Hay casi tantas posibilidades como tipos de electroforesis existen. Luego son transferidas a una membrana adsorbente para poder buscar la proteína de interés con anticuerpos específicos contra ella. Finalmente, se detecta la unión antígeno-anticuerpo por actividad enzimática, fluorescencia entre otros métodos. De esta forma se puede estudiar la presencia de la proteína en el extracto y analizar su cantidad relativa respecto a otras proteínas. Esta técnica es hoy en día imprescindible en varios campos de la biología, como la biología molecular, la bioquímica, la biotecnología o la inmunología. Existe toda una industria especializada en la venta de anticuerpos contra decenas de miles de proteínas diferentes.
El Western blot fue desarrollado en el laboratorio de George Stark, en la Universidad de Stanford. El nombre le fue dado por W. Neal Burnette,y consiste en un juego de palabras con una técnica análoga pero que usa DNA, el Southern blot, que en este caso debe su nombre a su descubridor, Edwin Southern. Otras técnicas que fueron nombradas siguiendo este criterio son el Northern blot, el Eastern blot, el Southwestern blot.
Biografía:
http://es.slideshare.net/tratamiento1/fundamento-y-procedimiento-de-las-pruebas-de-western-blot.http://www.abcam.com/index.html?pageconfig=resource&rid=11375&source=pagetrap&viapagetrap=westernblotguide.
Otras paginas relacionadas con esta información:
http://biologia.laguia2000.com/tecnicas-en-biologia/western-blot.
http://es.wikipedia.org/wiki/Southern_blot.
http://datateca.unad.edu.co/contenidos/203027/MODULO_Y_PROTOCOLO_EXE/leccin_11_la_tcnica_del_southern_blot.html.
para alguna aclaracion mas, visite este video:
http://youtu.be/VmCTsmhx2_w.
http://youtu.be/ikVsaRaFAYU
Jaime Moreno Lucena 2ºBHC
- X
- Xenopus
El Xenopus laevis o Rana Africana es un anuro de cuerpo robusto con la piel suave. Hay una salvaje y otra que se ha acostumbrado al cautiverio. En cuanto a las características, la salvaje tiene el dorso de un color marrón con manchas algo más oscuras y el vientre claro y, en cambio, la acostumbrada al cautiverio tiene el dorso color crema y el vientre blanco plateado. Ambas tienen el cuerpo plano, cabeza pequeña, manos y patas musculosas palmeadas con garras en las patas traseras. No tienen membrana timpánica. Cuando son adultas se especializan en la visión nocturna y tienen un gran desarrollo del olfato. Han desarrollado los ''órganos de la linea lateral'' que les permite captar estímulos en el agua. No tienen ni lengua ni dientes y las papilas gustativas se encuentran por casi toda la boca. En cuanto a diferencias entre hembras y machos tenemos que las hembras son más grandes y que cuando están maduras sexualmente tienen más prominentes los labios cloacales. Es una rana carnívora que se alimenta, sobretodo, de pequeños insectos que caen al agua.

Aquí podemos ver a la rana alimentarse de pequeños peces y alertarse por pequeños ruidos.
Los humanos la utilizamos para estudios de segmentación embrionaria puesto que sus embriones y ovocitos son muy fáciles de manipular.
En esta página podemos encontrar mucha más información sobre ella https://anfibios.paradais-sphynx.com/anuros/xenopus-laevis.htm
Bibliografía: Wikipediaorg. 2016. Wikipediaorg. [Online]. [5 October 2016]. Available from: https://es.wikipedia.org/wiki/Xenopus_laevis
Wikifauniacom. 2016. Wikifauniacom. [Online]. [5 October 2016]. Available from: http://wikifaunia.com/anfibios/xenopus-laevis/
Youtubecom. 2016. YouTube. [Online]. [5 October 2016]. Available from: https://www.youtube.com/watch?v=pdWaQVTGEJ0
Dicytcom. 2016. Dicytcom. [Online]. [5 October 2016]. Available from: http://www.dicyt.com/data/86/14986.jpg
Autora: Bárbara Pellejero
- Y
- Z